Search Thermo Fisher Scientific
Platinum SuperFi II DNA聚合酶─高保真PCR酶

產品亮點
- 卓越的準確度—經次世代定序鑑定,其保真度高於Taq保真度300倍以上。
- 簡化的工作流程—優化的緩衝液專為60°C的引子黏合溫度而配置(無需Tm計算器)。
- 更高的PCR成功率—可針對富含GC的目標物、純度不佳的DNA和長片段序列進行穩健的擴增。
- 實現自動化—透過Invitrogen Platinum熱啟動技術,可確保反應設置後24小時內的高特異性和室溫穩定性。
- 減少移液步驟—我們提供含、或不含直接凝膠上樣染料的預混液選項。
使用客戶對於Platinum SuperFi II聚合酶的評價
Platinum SuperFi II DNA聚合酶的優勢
>300x Taq高保真度
Platinum SuperFi II DNA聚合酶的錯誤率極低,在確保DNA序列準確性方面具有最高的可信度。使用次世代定序方法檢測,Platinum SuperFi II DNA聚合酶的相對保真度經計算後高於Taq DNA聚合酶的300倍。

圖1. 市售商用酶相對於Taq酶的保真度比較。 使用不同的DNA聚合酶,透過PCR擴增3.9 kb的序列,然後以MuA轉座酶將所得之PCR擴增子進行片段化。在片段化過程中引入了由12個隨機核苷酸組成的獨特分子標籤(Unique molecular identifiers,UMI),以單獨標記每一個產物。經次世代定序後,將讀數(reads)與正確序列進行比對,依照UMI家族分組,藉此發現擴增中的錯誤。只有當錯誤存在於UMI家族的所有reads中時,錯誤才得以識別;否則將被視為定序錯誤而被丟棄。以Taq DNA聚合酶作為標準來衡量聚合酶的保真度。請點擊此處,詳細閱讀研究文章。
影片:什麼是高保真PCR?
了解DNA聚合酶的保真度、測量酶保真度的方法,以及在您的PCR中使用高保真DNA聚合酶的優勢。
60°C的引子黏合溫度
Platinum SuperFi II緩衝液的獨特配方有助於減少PCR中繁瑣的優化步驟。使用Platinum SuperFi II DNA聚合酶時,不再需要計算黏合步驟的引子解鏈溫度。遵循常規引子設計原則(圖2)所得的產物,無論其序列如何,創新的緩衝液配方均能在60°C進行引子黏合。當在黏合步驟中使用計算出之Tms時,該緩衝液同樣能確保成功擴增(數據未顯示)。

圖2. Platinum SuperFi II DNA聚合酶在60°C的通用黏合溫度下可生成具有高特異性和高產率的PCR產物。使用不同黏合溫度的引子組,在60°C黏合溫度下從50 ng人類基因組DNA中擴增12個目標物。所使用的分子量標準參照物是Invitrogen TrackIt 1 Kb Plus DNA Ladder。上述用於Platinum SuperFi DNA聚合酶的黏合溫度是透過Tm計算器所得之。
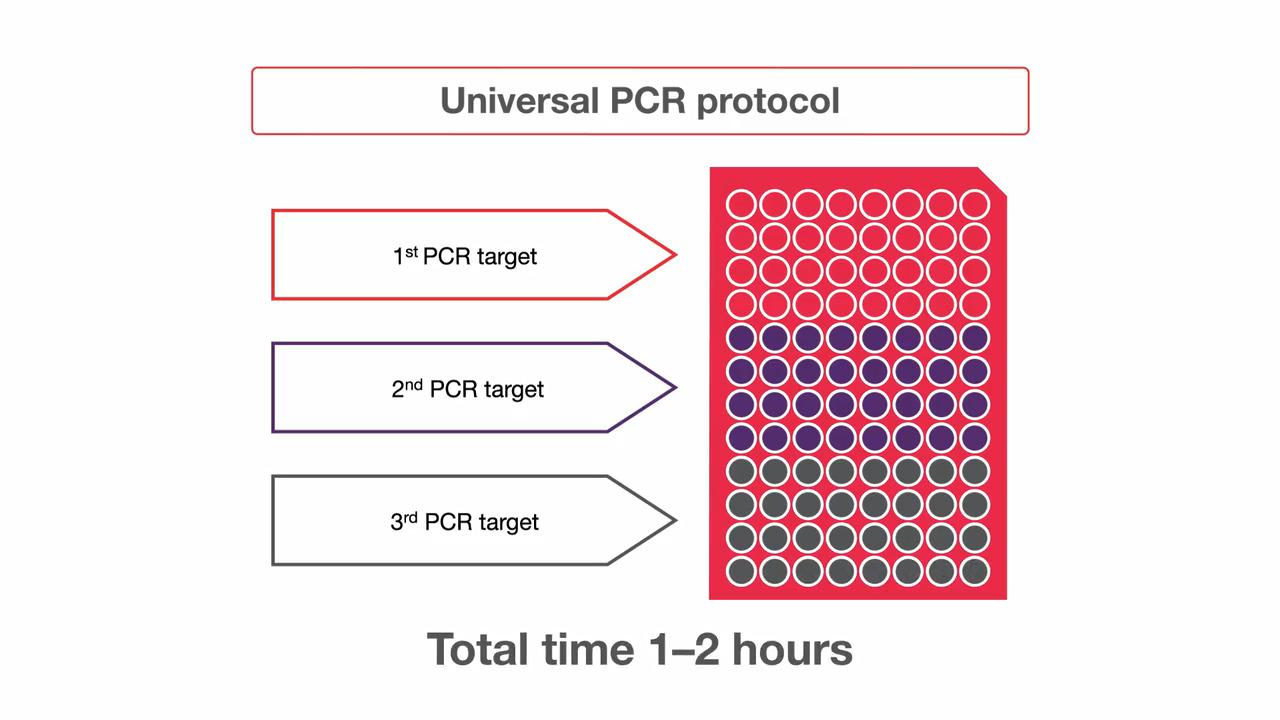
通用的PCR擴增方案
由於採用了創新型的反應緩衝液,Platinum SuperFi II DNA聚合酶擁有通用的黏合溫度和靈活的延長時間,從而實現所有PCR反應的共循環擴增(co-cycling)。

圖3. 透過通用PCR擴增方案,可節省時間並實現不同PCR反應的同時擴增。由於引子黏合溫度和延伸步驟不同,使用常規PCR試劑來進行PCR時,每個DNA片段都需要特定的擴增方案;因此,多個目標物通常無法在一次PCR運行中同時擴增。透過Platinum SuperFi II DNA聚合酶,因其具有通用的引子黏合溫度,可針對最長片段選擇擴增步驟時間,以實現在一個方案中同時進行不同的PCR擴增(圖4)。

圖4. Platinum SuperFi II DNA聚合酶,可實現短片段和長片段的同時擴增。從100 ng人類基因組DNA中擴增0.7 kb、2.0 kb、4.8 kb和14 kb的片段,均使用相同的擴增方案:98°C變性10秒,60°C黏合10秒,72°C延伸7分鐘。延伸時間是根據最長目標片段來進行計算。
直接凝膠上樣電泳
Platinum SuperFi II Green PCR Master Mix提供了PCR產物直接凝膠上樣的便利性,省去了向PCR樣品中添加染料的繁瑣步驟,並有助於減少移液錯誤。綠色緩衝液兼容於下游實驗應用,包括DNA定序、接合和限制性酶切。

圖5. 綠色預混液,可實現將PCR產物直接加樣至凝膠中上進行分析。Platinum SuperFi II DNA聚合酶的預混液,以綠色緩衝液規格提供,其中包含一種密度試劑和兩種追踪染料。使用這兩種染料(藍色和黃色),可以在電泳過程中輕鬆觀察DNA條帶的遷移(如右圖中電泳5分鐘和15分鐘的條帶)。
>300x Taq高保真度
Platinum SuperFi II DNA聚合酶的錯誤率極低,在確保DNA序列準確性方面具有最高的可信度。使用次世代定序方法檢測,Platinum SuperFi II DNA聚合酶的相對保真度經計算後高於Taq DNA聚合酶的300倍。

圖1. 市售商用酶相對於Taq酶的保真度比較。 使用不同的DNA聚合酶,透過PCR擴增3.9 kb的序列,然後以MuA轉座酶將所得之PCR擴增子進行片段化。在片段化過程中引入了由12個隨機核苷酸組成的獨特分子標籤(Unique molecular identifiers,UMI),以單獨標記每一個產物。經次世代定序後,將讀數(reads)與正確序列進行比對,依照UMI家族分組,藉此發現擴增中的錯誤。只有當錯誤存在於UMI家族的所有reads中時,錯誤才得以識別;否則將被視為定序錯誤而被丟棄。以Taq DNA聚合酶作為標準來衡量聚合酶的保真度。請點擊此處,詳細閱讀研究文章。
影片:什麼是高保真PCR?
了解DNA聚合酶的保真度、測量酶保真度的方法,以及在您的PCR中使用高保真DNA聚合酶的優勢。
60°C的引子黏合溫度
Platinum SuperFi II緩衝液的獨特配方有助於減少PCR中繁瑣的優化步驟。使用Platinum SuperFi II DNA聚合酶時,不再需要計算黏合步驟的引子解鏈溫度。遵循常規引子設計原則(圖2)所得的產物,無論其序列如何,創新的緩衝液配方均能在60°C進行引子黏合。當在黏合步驟中使用計算出之Tms時,該緩衝液同樣能確保成功擴增(數據未顯示)。

圖2. Platinum SuperFi II DNA聚合酶在60°C的通用黏合溫度下可生成具有高特異性和高產率的PCR產物。使用不同黏合溫度的引子組,在60°C黏合溫度下從50 ng人類基因組DNA中擴增12個目標物。所使用的分子量標準參照物是Invitrogen TrackIt 1 Kb Plus DNA Ladder。上述用於Platinum SuperFi DNA聚合酶的黏合溫度是透過Tm計算器所得之。
通用的PCR擴增方案
由於採用了創新型的反應緩衝液,Platinum SuperFi II DNA聚合酶擁有通用的黏合溫度和靈活的延長時間,從而實現所有PCR反應的共循環擴增(co-cycling)。

圖3. 透過通用PCR擴增方案,可節省時間並實現不同PCR反應的同時擴增。由於引子黏合溫度和延伸步驟不同,使用常規PCR試劑來進行PCR時,每個DNA片段都需要特定的擴增方案;因此,多個目標物通常無法在一次PCR運行中同時擴增。透過Platinum SuperFi II DNA聚合酶,因其具有通用的引子黏合溫度,可針對最長片段選擇擴增步驟時間,以實現在一個方案中同時進行不同的PCR擴增(圖4)。

圖4. Platinum SuperFi II DNA聚合酶,可實現短片段和長片段的同時擴增。從100 ng人類基因組DNA中擴增0.7 kb、2.0 kb、4.8 kb和14 kb的片段,均使用相同的擴增方案:98°C變性10秒,60°C黏合10秒,72°C延伸7分鐘。延伸時間是根據最長目標片段來進行計算。
直接凝膠上樣電泳
Platinum SuperFi II Green PCR Master Mix提供了PCR產物直接凝膠上樣的便利性,省去了向PCR樣品中添加染料的繁瑣步驟,並有助於減少移液錯誤。綠色緩衝液兼容於下游實驗應用,包括DNA定序、接合和限制性酶切。

圖5. 綠色預混液,可實現將PCR產物直接加樣至凝膠中上進行分析。Platinum SuperFi II DNA聚合酶的預混液,以綠色緩衝液規格提供,其中包含一種密度試劑和兩種追踪染料。使用這兩種染料(藍色和黃色),可以在電泳過程中輕鬆觀察DNA條帶的遷移(如右圖中電泳5分鐘和15分鐘的條帶)。
Platinum SuperFi II DNA聚合酶提供的穩健性
由於酶的穩健性和出色的熱啟動技術,Platinum SuperFi II DNA聚合酶可高特異性和高產量地擴增多種不同長度的目標物。基於抗體的Platinum熱啟動技術在最初的PCR變性步驟之前會抑制酶的活性,防止非特異性擴增和引子降解,同時提高目標擴增片段的產量。

圖6. 適用於擴增多種不同長度的寬範圍片段,且用途廣泛。Platinum SuperFi II DNA聚合酶(最左側)可高產量和高特異性地從100 ng人類基因組DNA擴增0.3 kb至14 kb範圍的DNA片段。同時使用其他供應商的DNA聚合酶,擴增相同的目標物:A—Merck KOD Hot Start,B—KAPA HiFi HotStart PCR試劑盒,C—PrimeSTAR GXL。所使用的分子量標準參照物為TrackIt 1 Kb Plus DNA Ladder。
Platinum SuperFi II DNA聚合酶的高靈敏度,可以檢測低豐度DNA模板並獲得準確的結果。在起始材料數量有限或樣品中目標DNA濃度較低的實驗中,高靈敏度聚合酶是非常有利的。

圖7. 從少量起始DNA中實現高靈敏度和可靠的擴增。Platinum SuperFi II DNA聚合酶(最左側)可分別從0.4 ng、2 ng、10 ng、50 ng和250 ng人類基因組DNA中可靠地擴增2 kb片段(模板量標示於每個泳道上方)。使用其他供應商的DNA聚合酶擴增相同的目標物:A—PfuUltra II Fusion Hot Start,B—HotStar HiFidelity DNA聚合酶試劑盒,C —Expand HiFi PLUS Enzyme Blend。預估的拷貝數為每0.4 ng人類基因組DNA約100個拷貝。所使用的分子量標準參照物為TrackIt 1 Kb Plus DNA Ladder。
Platinum SuperFi II DNA聚合酶於DNA結合結構域進行了改造設計,從而實現高持續合成能力,並提高對氯化血紅素(hemin,紅血球成分)、木聚醣(xylan,植物生物聚合物)和腐植酸(humic acid,存在於土壤)等常見PCR抑制劑的耐受性。

圖8. Platinum SuperFi II DNA聚合酶顯示對常見PCR抑制劑的高度耐受性。使用Platinum SuperFi II DNA聚合酶或其他供應商的高保真DNA聚合酶,從50 ng人類基因組DNA中擴增2 kb人類基因組DNA片段:A—Q5 Hot Start High-Fidelity,B—PrimeSTAR GXL,C—Merck KOD Hot Start,和D—KAPA HiFi HotStart PCR試劑盒,反應混合物中含有1—無抑制劑,2—腐植酸(4 µg/mL),3—血紅素(20 µM)或4—膽鹽(1毫克/毫升)。所使用的分子量標準參照物為TrackIt 1 Kb Plus DNA Ladder。
Platinum SuperFi II DNA聚合酶在室溫下具有更高的穩定性,可實現高通量應用。其卓越的Platinum熱啟動技術可實現室溫穩定性和酶的高特異性。

圖9. 使用Platinum SuperFi II DNA聚合酶配製的反應系統在室溫下十分穩定。從50 ng人類基因組DNA中擴增出0.5 kb片段。PCR反應分別在室溫下放置0小時和24小時,然後加載至Applied Biosystems ProFlex熱循環儀進行擴增。即使經過24小時的室溫設置,Platinum SuperFi II DNA聚合酶(lane P)也能獲得高特異性和高產量的擴增結果。同時使用其他供應商的DNA聚合酶進行相同的實驗:A—Q5 Hot Start High-Fidelity,B—KAPA HiFi HotStart PCR Kit,C—PrimeSTAR GXL。所使用的分子量標準參照物為TrackIt 1 Kb Plus DNA Ladder。
使用Platinum SuperFi II DNA聚合酶對不同類型的模板進行PCR擴增
Platinum SuperFi II DNA聚合酶憑藉其酶的穩健性及特別配製的專用緩衝液,能夠高特異性地擴增範圍寬廣的各種序列。Platinum SuperFi II緩衝液可以擴增富含GC的目標物,而無需補充DNA解鏈添加劑。

圖10. Platinum SuperFi II DNA聚合酶可對富含AT和GC的目標物進行穩健擴增。從50 ng人類基因組DNA中擴增出15個具有不同GC含量的目標物,且無需添加任何有助於DNA變性的補充緩衝液。所使用的分子量標準參照物為TrackIt 1 Kb Plus DNA Ladder。

圖11. 強勁擴增富含GC的目標物。Platinum SuperFi II DNA聚合酶可對富含GC的困難目標物(最左側)進行高特異性和高產量的擴增,而無需補充任何DNA解鏈添加劑。從50 ng人類基因組DNA中擴增出四個富含GC的片段(長度分別為0.74 kb、0.58 kb、0.71 kb和0.72kb;GC含量標示於上方)。根據製造商所推薦用於富含GC模板的PCR擴增方案,同時使用其他供應商的DNA聚合酶以擴增相同的目標物:A—PrimeSTAR GXL DNA聚合酶,B—KAPA HiFi HotStart PCR試劑盒(搭配專用於富含GC片段的特殊反應緩衝液),C—添加了10% DMSO的Merck KOD熱啟動DNA聚合酶。所使用的分子量標準參照物為TrackIt 1 Kb Plus DNA Ladder。
由於其高持續合成能力和極低的錯誤率,Platinum SuperFi II DNA聚合酶是準確擴增長片段(長達20 kb)的理想選擇,而且產量高、特異性極強。其還可以擴增更長的目標物片段(長達40 kb),但可能需要進行額外優化,如使用高品質模板(純淨、新鮮和完整)和新鮮的引子溶液。若是將引子濃度降低至0.2 μM也可以會改善PCR擴增結果。

圖12. 長片段的擴增。使用Platinum SuperFi II DNA聚合酶(lane P)成功從200 ng人類基因組DNA中擴增出20 kb的目標物。使用相同的引子組,並測試其他供應商的DNA聚合酶:A—Q5 Hot Start High-Fidelity,B—KAPA HiFi HotStart PCR Kit,C—Merck KOD Hot Start、D—PrimeSTAR GXL,和E—PfuUltra II Fusion HotStart。所使用的分子量標準參照物為TrackIt 1 Kb Plus DNA Ladder。

圖13. 擴增大於20 kb的目標片段。使用Platinum SuperFi II DNA聚合酶(lane P)成功從50 ng大腸桿菌基因組DNA中擴增出30 kb和40 kb的目標物。使用相同的引子組,並測試其他供應商的DNA聚合酶:A—Q5 Hot Start High-Fidelity,B—KAPA HiFi HotStart PCR Kit,C—Merck KOD Hot Start,D—PrimeSTAR GXL,和E—PfuUltra II Fusion HotStart。所使用的分子量標準參照物為TrackIt 1 Kb Plus DNA Ladder。

圖14. 使用Platinum SuperFi II DNA聚合酶在對不同濃度模板進行多重PCR擴增。從2.5 ng、25 ng和250 ng人類基因組DNA中(模板量標示於每個泳道上方),分別擴增出15個目標物(99 bp;131 bp;160 bp;199 bp;251 bp;300 bp;345 bp;400 bp;516 bp;613 bp;735 bp;908 bp;1,005 bp;1,190 bp和1,606 bp)。所使用的分子量標準參照物為TrackIt 100 bp DNA Ladder。
由於具有高特異性和高抑制劑耐受性,Platinum SuperFi II DNA聚合酶能夠高效擴增品質不佳的DNA模板,如福爾馬林固定的石蠟包埋(formalin fixed paraffin-embedded,FFPE)樣品。

圖15. 對從FFPE提取DNA樣品的擴增。透過Invitrogen RecoverAll FFPE總核酸分離試劑盒Invitrogen RecoverAll FFPE總核酸分離試劑盒所提取的10 ng小鼠FFPE DNA,使用Platinum SuperFi II DNA聚合酶可成功擴增長達0.4 kb的片段。所使用的分子量標準參照物為TrackIt 100 bp DNA Ladder。
Platinum SuperFi II DNA聚合酶和Platinum SuperFi DNA聚合酶之間的差異
Platinum SuperFi II DNA聚合酶的反應緩衝液,其配方專門添加了共穩定組分。這種獨特的緩衝液成分具有多種優勢:無需對引子進行Tm計算、可對富含GC的目標物進行更穩健的擴增、增強對長序列的擴增能力,以及適用於高通量PCR的通用PCR擴增方案(表1)。
表1. Platinum SuperFi II和Platinum SuperFi DNA聚合酶之間的比較。
| Platinum SuperFi II DNA聚合酶 | Platinum SuperFi DNA聚合酶 | |
|---|---|---|
| 保真度(與Taq相比) | 大於300x | 大於300x |
| 熱啟動修飾技術 | 是 | 是 |
| 需要Tm 計算器 | 否(引子的黏合溫度為60°C) | 是 |
| 通用PCR擴增方案 | 是 | 否 |
| 可擴增富含GC的模板 | 是(無需GC enhancer) | 是(建議使用GC enhancer) |
| 長片段擴增 | 長達20 kb(性能更強) | 長達20 kb |
| 室溫穩定性 | 可長達24小時 | 可長達24小時 |
| 對於抑制劑的耐受性 | 高 | 高 |
| 擴增子的 3′ 末端類型 | 平末端 | 平末端 |
| 經認證的低殘留DNA含量(每50 μL rxn) | ≤1個細菌 DNA拷貝 ≤0.3個人類DNA拷貝 | ≤1個細菌DNA拷貝 ≤0.2個人類DNA拷貝 |
Platinum SuperFi II和Platinum SuperFi DNA聚合酶的基準測試數據
References
Viral research
Other microbes
| Usage | Reference |
|---|---|
| Multiplex PCR for detection of S. aureus genes | Allam A, Fakhr A, Mahmoud M et al. (2021) Staphylococcus aureus nasal colonization among health care workers at an Egyptian tertiary care hospital. Microbes and Infectious Diseases 2(1):108–118. |
| PCR with gDNA and cDNA of fungal samples, followed by Sanger sequencing
| Ferrara M, Gallo A, Perrone G et al. (2020) Comparative genomic analysis of ochratoxin A biosynthetic cluster in producing fungi: new evidence of a cyclase gene involvement. Front Microbiol 11:581309. |
Metagenomics
| Usage | Reference |
|---|---|
| Amplification of bacterial full-length genes, followed by Sanger sequencing | Ferrara M, Haidukowski M, D'Imperio M et al. (2021) New insight into microbial degradation of mycotoxins during anaerobic digestion. Waste Manag 119:215–225. |
| Metabarcoding of fish DNA from pumped deep-sea water | Kawato M, Yoshida T, Miya M et al. (2021) Optimization of environmental DNA extraction and amplification methods for metabarcoding of deep-sea fish. MethodsX 8: 101238. |
| Rare mutation detection by molecular barcoding; fidelity measurement of different DNA polymerases | Mielinis P, Sukackaite R, Serapinaite A et al. (2021) MuA-based molecular indexing for rare mutation detection by next-generation sequencing. J Mol Bio 443 (19):167209. |
Immunology
| Usage | Reference |
|---|---|
| Cloning of VH and VL regions of an immunoglobulin | Granel J, Lemoine R, Morello E (2020) 4C3 human monoclonal antibody: a proof of concept for non-pathogenic proteinase 3 anti-neutrophil cytoplasmic antibodies in granulomatosis with polyangiitis. Front Immunol 11:573040. |
| RT-PCR with human liver and testis samples for microRNA expression analysis, followed by Sanger sequencing | Rubino E, Cruciani M, Tchitchek N (2021) Human ubiquitin-specific peptidase 18 is regulated by microRNAs via the 3'untranslated region, a sequence duplicated in long intergenic non-coding RNA genes residing in chr22q11.21. Front Genet 11:627007. |
Additional areas
Viral research
Other microbes
| Usage | Reference |
|---|---|
| Multiplex PCR for detection of S. aureus genes | Allam A, Fakhr A, Mahmoud M et al. (2021) Staphylococcus aureus nasal colonization among health care workers at an Egyptian tertiary care hospital. Microbes and Infectious Diseases 2(1):108–118. |
| PCR with gDNA and cDNA of fungal samples, followed by Sanger sequencing
| Ferrara M, Gallo A, Perrone G et al. (2020) Comparative genomic analysis of ochratoxin A biosynthetic cluster in producing fungi: new evidence of a cyclase gene involvement. Front Microbiol 11:581309. |
Metagenomics
| Usage | Reference |
|---|---|
| Amplification of bacterial full-length genes, followed by Sanger sequencing | Ferrara M, Haidukowski M, D'Imperio M et al. (2021) New insight into microbial degradation of mycotoxins during anaerobic digestion. Waste Manag 119:215–225. |
| Metabarcoding of fish DNA from pumped deep-sea water | Kawato M, Yoshida T, Miya M et al. (2021) Optimization of environmental DNA extraction and amplification methods for metabarcoding of deep-sea fish. MethodsX 8: 101238. |
| Rare mutation detection by molecular barcoding; fidelity measurement of different DNA polymerases | Mielinis P, Sukackaite R, Serapinaite A et al. (2021) MuA-based molecular indexing for rare mutation detection by next-generation sequencing. J Mol Bio 443 (19):167209. |
Immunology
| Usage | Reference |
|---|---|
| Cloning of VH and VL regions of an immunoglobulin | Granel J, Lemoine R, Morello E (2020) 4C3 human monoclonal antibody: a proof of concept for non-pathogenic proteinase 3 anti-neutrophil cytoplasmic antibodies in granulomatosis with polyangiitis. Front Immunol 11:573040. |
| RT-PCR with human liver and testis samples for microRNA expression analysis, followed by Sanger sequencing | Rubino E, Cruciani M, Tchitchek N (2021) Human ubiquitin-specific peptidase 18 is regulated by microRNAs via the 3'untranslated region, a sequence duplicated in long intergenic non-coding RNA genes residing in chr22q11.21. Front Genet 11:627007. |
Additional areas
訂購資訊
宣傳頁
使用手冊/方案
應用指南
白皮書
焦點文章
PCR教學中心
了解PCR的歷史、反應設置注意事項、DNA聚合酶選擇的重要性、常見方法和應用,以及疑難解答。
PCR和cDNA合成支援中心
查找適合您的終點PCR和cDNA應用的相關提示、疑難解答幫助和實用資源。
OEM和客製化PCR解決方案
根據您的特定要求,探索客製化的生產解決方案、產品標籤和包裝功能。
聯繫我們
透過電子郵件或電話聯繫我們的技術應用科學家,了解有關PCR酶和預混液的其他問題。
除非另有說明,所有商標均屬於Thermo Fisher Scientific及其子公司。Q5屬於New England BioLabs Inc.的商標。PrimeSTAR屬於Takara-Clontech Laboratories, Inc. 的商標。Merck屬於Merck KGaA的商標。KAPA HiFi HotStart屬於Roche Inc. 的商標。HotStar屬於Qiagen GmbH 的商標。Roche Expand屬於Roche Molecular Systems Inc. 的商標。PfuUltra屬於Agilent, Inc. 的商標。
僅供研究用途。不得用於診斷程序。