Search Thermo Fisher Scientific
TALEN 기술 정보
TALEN 또는 TAL 효과기는 생체 세포에서 정밀하고 효율적으로 유전자를 편집하는 데 널리 사용되는 기술입니다. 이 유전체 편집 기술은 박테리아, 효모, 식물, 곤충, 제브라피시 및 포유류를 포함한 다양한 숙주에서 기능하는 것으로 알려져 있습니다. 당사는 고객에게 TALEN을 사용한 연구의 시작과 지속적인 연구 개선에 대한 확신을 제공하기 위한 다양한 리소스 컬렉션을 보유하고 있습니다.
TALEN이란?
전사 활성화 인자류(Transcription activator-like, TAL) 효과기 단백질은 널리 분포된 식물 병원체인 Xanthomonas의 박테리아에 의해 생성됩니다. 천연 TAL은 숙주 DNA의 특정 염기서열에 결합하여, 감염된 식물의 유전자 발현을 질병을 더욱 진행시키는 방식으로 변화를 유발합니다. 천연 TAL 효과기 단백질에는 효과기 영역 및 매우 특이적인 DNA 결합 영역이라는 두 가지 영역이 있습니다.
DNA 결합 영역의 구조를 조작하여 유전체의 임의의 DNA 염기서열에 특별히 결합하는 단백질 영역을 생성할 수 있습니다. 그런 다음 특별히 변형된 DNA 결합 단백질 영역을 custom effector domain(예: nuclease 또는 전사 활성화 인자 또는 억제제)에 연결하여 정확하게 타겟 DNA 조작을 수행할 수 있는 키메라 단백질을 생성할 수 있습니다.
유전체 공학을 위한 타겟 nuclease를 통하거나 정밀 유도된 유전자 발현 조정자에 의해 연구자가 설계한 TALEN 효과기 단백질은 이미 세포, 분자, 합성 생물학을 비롯한 광범위한 과학 응용 분야와 약품 개발, 바이오 연료 연구 등의 발전을 돕고 있습니다.
TALEN은 어떻게 작동합니까?
Xanthomonas TAL 효과기에서 유래한 TAL의 DNA 결합 영역은 다양한 아미노산 반복 구간으로 구성됩니다. 각 반복 구간에는 33–35 아미노산이 포함되며 단일 DNA 염기 쌍을 인식합니다. DNA 인식은 그림 1과 같이 반복 가변 이잔기(Repeat-Variable Di-residue, RVD)라고 하는 각 반복 내의 12 및 13 위치에서 두 개의 초가변(hypervariable) 아미노산 잔기를 통해 이루어집니다.
TAL 효과기 반복은 모듈식으로 조립할 수 있으며, RVD를 조절하여 특정 타겟 DNA 염기서열을 인식하는 TAL 단백질을 생성할 수 있습니다. 반복 연결은 간단합니다. 긴 TAL 효과기를 특별히 유전체의 임의의 자리를 표적으로 하도록 설계할 수 있습니다.

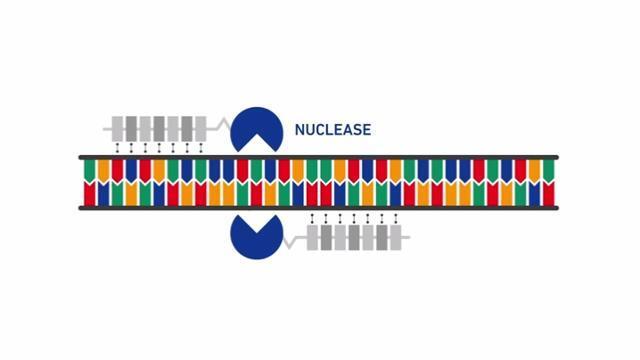
Fok1 nuclease와 융합된 GeneArt TAL 단백질 쌍을 사용하여 이중 가닥 DNA 절단을 지정된 유전체 위치에 생성할 수 있습니다(그림 2). 타겟 설정에 한 쌍의 TAL 단백질을 사용하여 off-target effect를 줄입니다.
Fok1 nuclease 영역에 의해 유도되는 절단은 이후 두 가지 내인성 세포 메커니즘, 즉 비상동성 말단 연결(nonhomologous end joining, NHEJ) 또는 상동성 유도 복구(homology-directed repair, HDR) 중 하나를 통해 복구됩니다.
- NHEJ는 오류가 발생하기 쉽고 단백질 코딩 유전자의 코딩 시퀀스 내에서 발생할 경우 종종 프레임 시프트 돌연변이를 유도하여 유전자를 효과적으로 발현하지 않도록 합니다.
- HDR에서 상동 DNA "donor 염기서열”을 사용하여 정의된 새로운 DNA 염기서열을 유도할 수 있습니다.
결과적으로 Fok1 endonuclease 융합된 단백질은 유전자가 발현하지 않도록 유도하거나 유전체 내 정확한 위치에 조작된 DNA 단편을 정확하게 삽입하는 데 사용할 수 있습니다.
TALEN 기술에 대해 자세히 알아보기

애플리케이션 노트: Sanger 염기서열 분석을 사용한 CRISPR 및 TALEN 매개 유전체 편집 워크플로우
유전체 편집 워크플로우에서 모세관 전기영동 및 Minor Variant Finder Software를 통한 생어 염기서열 분석이 어떻게 사용되는지 확인해 보십시오. CRISPR 매개 편집 결과가 제시되어 있지만 여기서 적용되는 원리는 ZFN 또는 TALEN 매개 편집 워크플로우에도 사용할 수 있습니다. 워크플로우의 단순함과 비용 효율성 그리고 복잡하지 않은 데이터 분석 덕분에 모세관 전기영동을 이용한 sanger 염기서열 분석은 유전체 편집 워크플로우의 중요한 부분으로 활용됩니다.

블로그: Straight From The Scientist—Jon Chesnut on CRISPR versus TALEN
CRISPR 및 TALEN 도구 및 기술은 현재 그리고 향후의 유전자 편집 방식을 바꾸고 있습니다. 이 두 가지 방법의 차이는 무엇입니까? 그리고 이 두 기술은 언제 사용하는 것이 좋습니까? 이 문서에서는 이러한 질문 등에 대해 설명합니다.

유전체 편집 리소스 라이브러리
유전체 편집에 대한 과학 애플리케이션 노트, 출판물, 동영상, 웨비나 및 과학 포스터 등의 전문 컬렉션을 이용할 수 있습니다.

유전체 편집 리소스 가이드
유전체 편집에 대한 소개 자료로 제공되는 이 리소스 가이드는 유전자 편집에 사용되는 다양한 기술을 비롯하여 편집된 세포의 설계, 전달 및 검출 방법에 대해 다룹니다.
TALEN 기술에 대한 추가 리소스 및 지원
For Research Use Only. Not for use in diagnostic procedures.
For Research Use Only. Not for use in diagnostic procedures.