Search Thermo Fisher Scientific

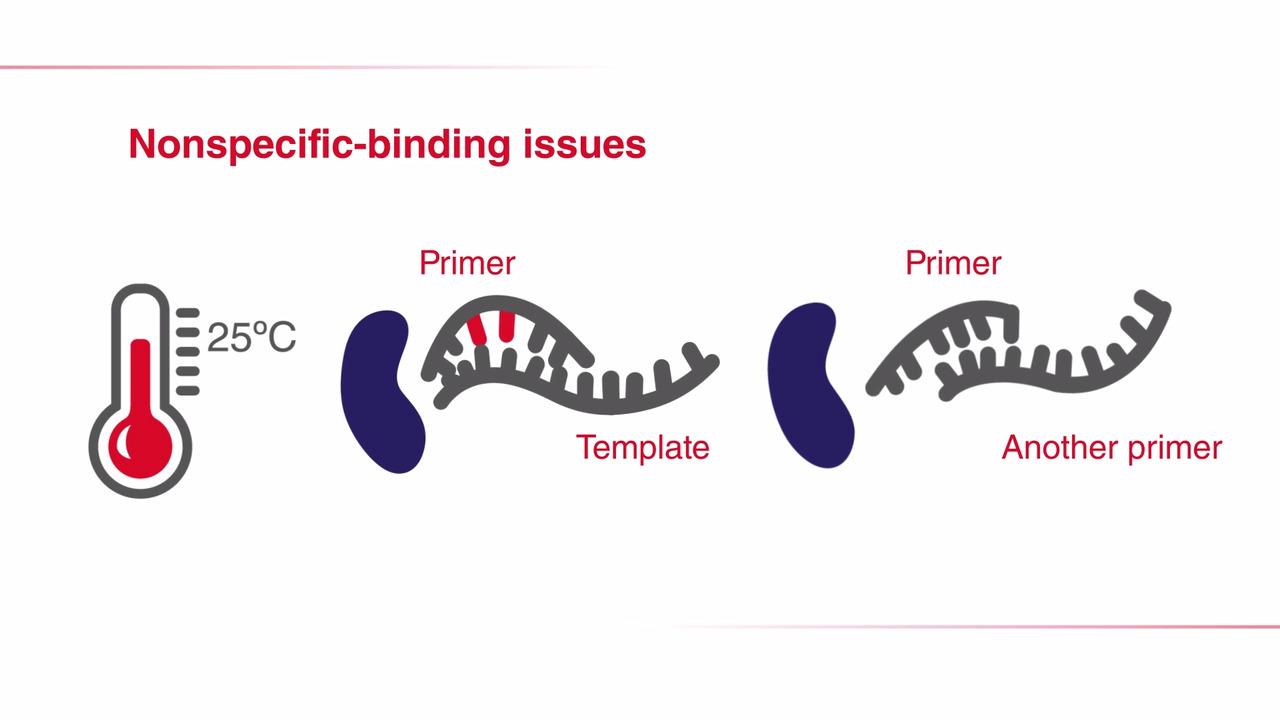
非特异性扩增是PCR反应最主要的障碍之一,因为它会显著降低目的基因扩增的产率和灵敏度,从而影响扩增结果的合理解释和下游实验应用的成功。而DNA聚合酶经常会延伸错配的目的基因和引物二聚体,这是非特异性扩增的常见来源。如何减少非特异性扩增?方案之一是在冰上配制PCR反应。这样有助于将DNA聚合酶的活性保持在低水平,但在PCR开始之前依然可能会合成不需要的产物。另一个办法是将DNA聚合酶的加入时间推迟到第一个循环的退火步骤。因为只有在90℃以上的初始变性步骤之后才能开始扩增,所以该技术被称为“热启动”。
了解热启动PCR及其对PCR应用的好处。了解如何在PCR中减少非特异性扩增并提高产量。
尽管手动热启动程序能够有效改善特异性,但是其过程不仅费时费力,而且会增加样品污染和降低可重复性的风险。1994年,具有真正热启动特性的TaqDNA聚合酶问世[1,2],通过在室温下配制反应时将特异性抗体与聚合酶结合来抑制其活性。在初始高温变性步骤(如,>90℃)阶段,结合的抗体失活,从而激活DNA聚合酶(图1)。
图 1.基于抗体的热启动DNA聚合酶及其在PCR反应体系中的激活提高了特异性。
此外,变性步骤还可能分离反应配制过程中形成的错配序列和引物二聚体,从而防止它们在后续退火和延伸过程中被DNA聚合酶扩增。这样,热启动DNA聚合酶便可降低非特异性扩增,提高产率,可实现室温下配制反应体系,方便用于高通量实验应用(图2-4)。
图 3.热启动DNA聚合酶适用于室温下配制反应体系,可用于高通量应用。准备PCR反应并在室温下孵育0、24和72小时, 然后放置到热循环仪中。即使在室温配制反应体系72小时后依然能够高特异性地扩增来自人类gDNA的2 kb片段,表明热启动DNA聚合酶可适用于大规模实验。
图 4.聚合酶活性对比:(A)真正的“热启动”DNA聚合酶和(B) “暖启动”DNA聚合酶。在60°C(恒温)下检测60分钟内的聚合酶活性。在热激活测试(蓝色曲线)中,在 94°C下对聚合酶加热处理2分钟,使抗体与聚合酶解离。不使用热激活(红色曲线)时,真正的热启动DNA聚合酶无法检测到聚合酶活性,而暖启动酶可在60°C被激活,因此不适用于热启动实验应用。
作为抗体的替代物,通过对酶活性位点进行热不稳定化学修饰,或者使用适配子等小分子缩短活化时间,也能够获得热启动性能。无论是否选择热启动技术,在非加热条件下有效阻断DNA聚合酶活性,对确保特异性而言都至关重要。(图4)
使用Invitrogen Platinum II Taq 热启动DNA聚合酶,在高通量应用中,可以在反应设置后保持24小时室温稳定,并同时扩增不同的PCR靶标。
由于热循环是DNA扩增重复链式反应条件的关键特征,因此,所使用DNA聚合酶的热稳定性非常重要。尽管最初来自于嗜热菌菌株的 Taq DNA 聚合酶可耐受相对较高的温度,但是其半衰期在90°C以上时明显缩短。当使用 长时间高温 使具有二级结构和富含GC序列的DNA变性时,这一缺陷便成为一大难题。同样,在扩增长片段模板时,需要更大量的Taq DNA 聚合酶或补充Taq DNA聚合酶,以供长时间孵育。因此,从超嗜热菌分离的DNA聚合酶具有更高的热稳定性,将有助于克服这些挑战。
Pfu DNA聚合酶是一种为大家所熟知的超耐热酶,来自于水热环境中的超嗜热古细菌 Pyrococcus furiosus 。在95°C下,Pfu聚合酶的稳定性能比 Taq 聚合酶高20倍[3]。其它常见的超耐热DNA聚合酶包括来自古细菌 Thermococcus 和 Pyrococcus 的KOD和GBD等。
尽管古细菌DNA聚合酶的热稳定性极强,但它们在某些方面也具有一定局限性。例如,超耐热 Pfu DNA 聚合酶的合成能力较低(与 Taq DNA聚合酶相比),因此合成DNA的速度较慢。此外,古细菌DNA聚合酶无法扩增含有尿嘧啶的DNA模板,因为其存在尿嘧啶结合区域作为一种DNA修复机制[4,5]。含尿嘧啶的DNA序列是 防止PCR 残余污染 和经亚硫酸氢盐转化进行 基因座甲基化分析 的基础。
DNA聚合酶的校正能力决定了保真度,会影响DNA序列复制的准确性。因此,高保真DNA聚合酶就是具有强校正活性的酶。DNA聚合酶准确复制DNA序列(即获得无错误序列)的能力对分子克隆、测序和定点突变等实验应用至关重要(应用指南:定点突变)。
了解DNA聚合酶的保真度,测量酶保真度的方法,以及在您的PCR中使用高保真DNA聚合酶的好处。
其校正活性基于其 3′ → 5′核酸外切酶活性,可校正错误插入的核苷酸。DNA聚合酶上的核酸外切酶活性位点与其5′→ 3′聚合酶活性位点是分离的(图5)。当错配的核苷酸插入聚合结构域,DNA合成将因不合适的碱基配对动力学而暂停。暂停期间,DNA聚合酶将切除错配的核苷酸并用正确的核苷酸替换[6]。
图 5.DNA聚合酶及其 5′→3′聚合酶结构域和3′→5′核酸外切酶结构域(基于 大肠杆菌 DNA聚合酶I的结构)。
DNA聚合酶的保真度检测方法多种多样,如菌落筛选测定、Sanger测序和下一代测序[7-10]。传统的菌落筛选方法,是将PCR扩增片段( lac 基因的)克隆到质粒中。使用 蓝白斑筛选检测重组质粒转化的菌落。lac 基因插入片段上的突变(可能由于PCR过程中的错误复制产生)导致lacZ功能丧失,形成白色菌落。另一方面,无PCR错误的 lac 插入片段则形成蓝色菌落。使用Sanger测序对克隆的PCR片段进行测序,可检测DNA聚合酶的错误率。利用下一代测序,可直接对PCR扩增子进行测序(图 6)。
图 6.检测DNA聚合酶保真度的常用方法。
DNA聚合酶的保真度常用错误率的倒数表示(保真度=1/错误率),指发生错配的核苷酸数与聚合的总核苷酸数的比值。因此,DNA聚合酶保真度高度依赖于PCR扩增子长度和PCR扩增循环数。为了准确对比不同聚合酶的保真度,必须使用相同的方法和循环参数进行检测。
通常,保真度表示为与 Taq DNA 聚合酶保真度的相对值。天然存在的校正DNA聚合酶(如 Pfu 和KOD)的保真度约为 Taq聚合酶的10倍。但是,“下一代”高保真DNA聚合酶经过定向改造,具有超高的保真度, >约为 Taq 聚合酶的50-100倍(图7)。使用这些酶,错配率可低至几百万分之一。
图 7.利用下一代测序方法,对比常用的高保真酶。
酶的合成能力可定义为在一次结合中处理的核苷酸数量。DNA聚合酶的合成能力通常反应了合成率和合成速度,以及酶与底物的亲和力。合成能力高的DNA聚合酶适用于扩增长模板、具有二级结构和富含GC的序列,以及存在肝素、木聚糖和腐殖酸等PCR抑制剂的血液和植物组织样品(图8)。
图 8.具有高合成能力的DNA聚合酶能够有效扩增(A)来自人类gDNA的不同长度目标序列,(B)各种GC含量(无增强子)的模板以及(C)来自含有常见PCR抑制剂的样品的目的片段。这些实验使用了具有高合成能力的 Invitrogen™ Platinum™ SuperFi™ DNA 聚合酶 。
使用Invitrogen Platinum II Taq DNA聚合酶,成功扩增纯度较低的DNA样本和富含GC的目标序列。
早期的高保真DNA聚合酶具有较强的核酸外切酶活性,所以合成能力较低,且会减慢聚合速度。因此,其扩增较长目标DNA的速度明显减慢。例如,校正 Pfu DNA聚合酶的保真度是 Taq DNA聚合酶的7倍,但其合成率还不到 Taq聚合酶的一半。使用另一个蛋白质的强DNA结合域对DNA聚合酶进行改造后,实现了合成能力的突破,同时不影响聚合酶活性(图9)[10]。这些改进型DNA聚合酶的合成能力提高了2-5倍。
图 9.DNA聚合酶的合成能力。(A)高合成能力的DNA聚合酶与其底物的亲和力更高,每次结合能引入更多核苷酸。(B)含有DNA结合域的DNA聚合酶序列示意图(Pol=聚合酶结构域, 3-′5′ exo = 3′→ 5′核酸外切酶结构域,DBD=DNA结合域,N=N末端,C=C末端)。
总之,DNA聚合酶的四大特点——特异性、热稳定性、保真度和合成能力——使这些酶具有多种功能,进一步拓展了它们在PCR中的应用。酶的 特异性 可确保获得高产率的目标PCR产物,同时尽量减少在克隆和定量等下游实验应用中的潜在问题。高 合成能力 和超耐热性 克服了扩增二级结构、富含GC的序列以及长DNA片段的困难。此外,提高 合成能力 使酶能够耐受DNA样品中天然存在的PCR抑制剂。最后,高 保真性 提高了序列复制的准确性。
- Sharkey DJ, Scalice ER, Christy KG Jr (1994) Antibodies as thermolabile switches: high temperature triggering for the polymerase chain reaction.Biotechnology (N Y)12(5):506–509.
- Kellogg DE, Rybalkin I, Chen S (1994) TaqStart Antibody: "hot start"PCR facilitated by a neutralizing monoclonal antibody directed against Taq DNA polymerase.Biotechniques16(6):1134–1137.
- Pelt-Verkuil EV, Belkum AV, Hays JP (2008) Principles and Technical Aspects of PCR Amplification.Dordrecht: Springer.
- Lasken RS, Schuster DM, Rashtchian A (1996) Archaebacterial DNA polymerases tightly bind uracil-containing DNA.J Biol Chem271(30): 17692–17696.
- Fogg MJ, Pearl LH, Connolly BA (2002) Structural basis for uracil recognition by archaeal family B DNA polymerases.Nat Struct Biol9(12):922–927.
- Steitz TA (1999) DNA polymerases: structural diversity and common mechanisms.J Biol Chem274(25):17395–17398.
- Lundberg KS, Shoemaker DD, Adams MW et al.(1991) High-fidelity amplification using a thermostable DNA polymerase isolated from Pyrococcus furiosus.Gene108(1):1–6.
- Barnes WM (1992) The fidelity of Taq polymerase catalyzing PCR is improved by an N-terminal deletion.Gene112(1):29–35.
- Vandenbroucke 1, Van Marck H, Verhasselt P (2011) Minor variant detection in amplicons using 454 massive parallel pyrosequencing: experiences and considerations for successful applications.Biotechniques.51(3):167–177.
- Kinde I, Wu J, Papadopoulos N et al.Proc Natl Acad Sci U S A.108(23):9530–9535.
- Wang Y, Prosen DE, Mei L et al.(2004) A novel strategy to engineer DNA polymerases for enhanced processivity and improved performance in vitro.Nucleic Acids Res32(3):1197–1207.
仅供科研使用,不可用于诊断目的。