Search Thermo Fisher Scientific
ボリューム電子顕微鏡法でシリアルブロックフェイスイメージングが使用される理由
細胞や組織の複雑な3次元構造の解明を、天然状態のまま実施することは、生命システムにおける構造と機能の相互関係を知るうえで非常に重要です。近年、SEMをベースとした大容量組織の3D再構成のメソッドが大きく進歩しています。シリアルブロックフェイスSEM(SBF-SEM)では、プラスチック包埋された生体組織ブロックのイメージングとin situ連続スライスをSEM真空チャンバー内で組み合わせることで、大容量組織の再構築を完全に自動化しています。従来、軸方向の分解能は、ブロックフェイスから切断できる切片の最小厚さによって制限されていましたが、Thermo Scientific VolumeScope 2 SEMではSBF-SEMとマルチエネルギーデコンボリューションSEM(MED-SEM)を組み合わせることで、真の意味での等方的な3D分解能を持つ大容量イメージングが可能になりました。
シリアルブロックフェイス顕微鏡法の仕組み
シリアルブロックフェイスイメージングでは、まず樹脂包埋された組織サンプルの表面を電子ビームでスキャンして、標本の2D画像を取得します。
次に、この一番上の表面をin situミクロトームによって除去します。各切片をどの程度の厚さとするかはユーザー定義できますが、通常は15~20 µmより大きくします。この切片は廃棄され、デブリ収集装置によって集められます。
次に、新しい表面の画像がSEMで収集されます。このプロセスは、サンプル全体のイメージングが終了するまで繰り返され、最終的なサンプルの高さは数十から数百マイクロメートル以上に及ぶ場合もあります。その後、画像のシリアルスタックを、3Dレンダリングソフトウェアを使用して処理します。
シリアルブロックフェイスイメージングは最適化が可能です。特定のユーザーやサンプルの要件などのニーズに合わせたり、局所的な関心領域、複数のエリア、各種の画像検出器を含めるよう調整したりすることができます。
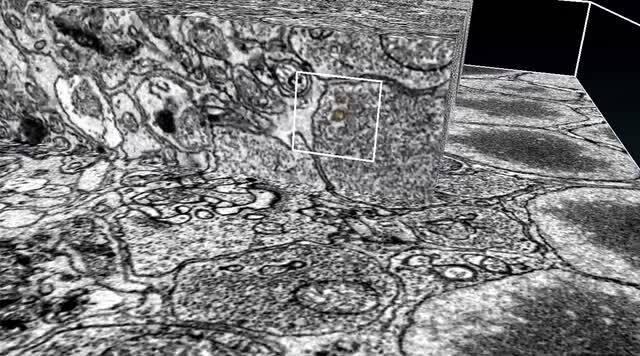
マルチエネルギーデコンボリューションSEM(MED-SEM)は新しい手法の1つで、機械的切片作製と光学的切片作製を組み合わせることで軸方向の分解能を向上させます。ダイヤモンド刃を使用してin situでブロックから切片を切り出した後、露出した表面のSEMイメージングを複数の加速電圧設定で行います。その後、これらの画像にデコンボリューションアルゴリズムを適用することで、表面下の光学層を何枚か生成し、それらを用いて3次元サブセットを構築します。VolumeScope 2 SEMではこのサイクルを繰り返すことで、Z分解能10 nmの等方性データセットを取得します。
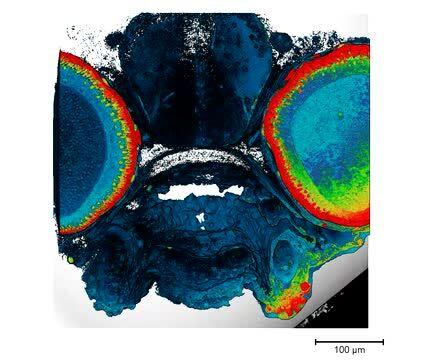
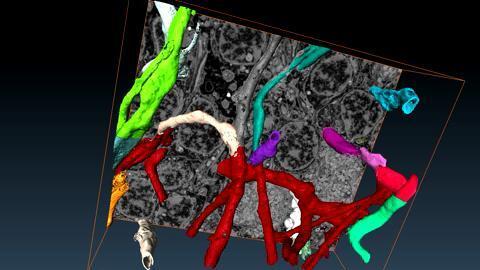
シリアルブロックフェイスSEMの3Dサンプルギャラリー
ミトコンドリアの超微細構造
可視化ソフトウェアとしてThermo Scientific Amiraを用いる標準化されたプロトコルによってオルガネラ形態を3Dで定量化。これにより、これらの細胞下部構造の測定における正確性と再現性を確保。プロトコルの適用にSBF-SEMとAmiraソフトウェアを用いることで、ミトコンドリアと小胞体(ER)の構造を定量化。

シリアルブロックフェイス走査型電子顕微鏡法を使用している研究者
「かつてはネットサーフィンをしていましたが、今はデータセットをサーフィンしています。構造とは機能であり、それが細胞生物学のドグマです。」
メイヨークリニックのJeffrey Salisbury教授が、45年以上の顕微鏡使用経験について論じています。同氏は、最近の3D研究を含むさまざまな生物学的疑問を解明するため、正常時と疾患状態の組織、腎臓、脳のさまざまな標本の3D構造を、シリアルブロックフェイスイメージングを用いて観察してきました。
「本物の細胞生物学を行う機会が到来しました。何年もの間それを待望していた私にとって、その開始は待ちきれません。」
パデュー電子顕微鏡センター所長のChristopher Gilpin氏が、シリアルブロックフェイスイメージング、およびその3Dイメージングテクニックでの生物学的マルチスケールマッピングにおける潜在的な用途について論じています。
シリアルブロックフェイスイメージングの関連リソース
For Research Use Only. Not for use in diagnostic procedures.