Search Thermo Fisher Scientific
逆転写反応のセットアップ:重要な7つの注意点

逆転写反応のセットアップ
分子生物学実験において逆転写は主に、組織や細胞に特異的に発現しているRNAを鋳型にして相補的DNA(cDNA)を作製するために行われます。そのような実験を成功させるためには、テンプレート、試薬類、反応条件に関して、重要な注意点があります。
このページの内容:
RNAテンプレートの調製
RNAは逆転写においてテンプレートの役目をします。トータルRNAは日常的にRT-(q)PCRなどのダウンストリームアプリケーションのためのcDNA合成に使用されますが、一方で、特殊なタイプのRNA(例えば、メッセンジャーRNAmRNA)やmiRNAのようなスモールRNAなど)は、cDNAライブラリの構築やmiRNAプロファイリングを行う際に、濃縮が必要となる場合があります。
RNAをインタクトに保つことが極めて重要であり、抽出、調製、保存、実験での使用においては、特別な注意が必要となります。RNAの分解を防ぐための最善策は、グローブを着用し、エアロゾルバリアチップでピペット操作を行い、ヌクレアーゼフリーの実験器具と試薬類を使用し、実験エリアを汚染除去することです。
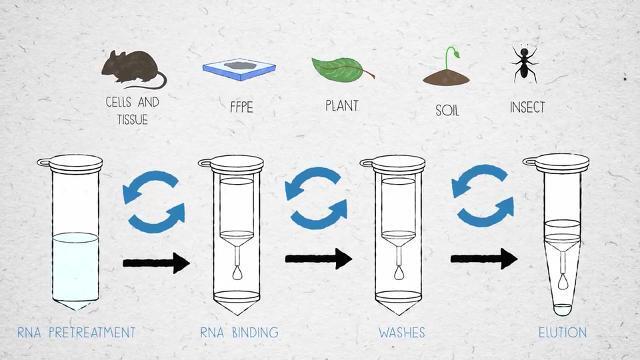
RNAの分離精製には、材料の種類(例えば、血液、組織、細胞、植物など)や実験の目的に応じて、様々な方法が利用できます。分離精製プロセスにおける主な目的は、適切な保存と抽出方法に基づいたRNA分子の安定化、RNasesの阻害、そして収量の最大化です。最適な精製法では、酵素活性に影響を及ぼす植物組織由来の複合多糖類やフミン酸、そして逆転写酵素の共通の阻害剤となる塩類や金属イオン、エタノール、フェノールのような内因性の化合物を除去します。一度精製したRNAは–80°Cで保存し、できるだけ凍結融解を繰り返さないようにします。
精製後のRNAの品質と収量を評価する方法が幾つかあります。一般的な方法は、特異的な波長における吸光度を測定するUV分光法です。RNAの収量はランベルト・ベールの法則に従って260 nmにおける吸光度から求めることができ、夾雑物の有無は異なる波長における吸光度の比率から推測できます(表 1)。全ての核酸類が似たような波長のUVを吸収するため、UV吸収がRNAに特異的ではないことに注意してください。より特異的で高感度な分析が必要な場合には、ターゲット分子と特異的に結合した時にだけシグナルを発生する色素を用いる蛍光アッセイが良いかもしれません。
表 1.RNA分析のためのUV測定ガイドライン
| 吸光度 | 存在が示唆されるもの | 標的となる比率 |
|---|---|---|
| 230 nm | 有機化合物、糖類、尿素、塩類 | A260/A230>1.8 |
| 260 nm | 全ての核酸類 | A260≈ 0.1~1.0 |
| 270 nm | フェノール | A260/ A270>1.0 |
| 280 nm | タンパク質 | RNA:A260/A280≈ 2.0 DNA:A260/A280≈ 1.8 |
| 330 nm | 光散乱 | A330= 0 |
RNAの品質は、28Sおよび18SリボソームRNA(rRNA)をトータルRNAとして比較することで評価できます。トータルRNAを変性後、ゲル電気泳動によってサイズごとに分離し、定性的に評価します。28S rRNAと18S rRNAのバンド強度を比較し、2:1となっていれば、それはRNAが分解していないことを意味します(図1A)。RNAの品質をより定量的に評価する方法としては、マイクロフルイディクスと特有のアルゴリズムを組み合わせた、アジレント・テクノロジー社の開発した方法が利用できます。この方法ではRNA Integrity Number、すなわちRINと呼ばれる数値表示が得られ、その値が8から10の間であれば、それはRNAが高品質であることを意味します[1,2] (図1B)。

ゲノムDNAの除去
場合によっては、微量のゲノムDNA(gDNA)がRNAと一緒に混ざって精製されることがあります。混入したgDNAは逆転写反応の妨げになり、擬陽性、高バックグラウンド、あるいはRT-qPCRにおける感度の低下につながる可能性があります。
一般的には、gDNAを除去するために、DNase Iを精製RNAに処理します。加えたDNase Iは、プライマーや合成したcDNAのような1本鎖DNAを分解してしまうため、RT-PCRの前に、完全に除去しておく必要があります。DNase Iの不活化(例えばEDTA処理や熱処理)や、酵素の除去といった手順は、しばしばRNAの分解やサンプルの損失をもたらします。
DNase Iの代替品として、RNAや1本鎖DNAに影響のない2本鎖特異的DNaseが、混入したgDNAの除去に利用できます。2本鎖特異的DNaseは熱に不安定な性質を持つため、悪影響のない比較的マイルドな温度(例えば55°Cなど)で、簡単に不活化できます。このような2本鎖特異的かつ熱不安定性のDNaseは、逆転写反応に先立ち、精製RNAと37°Cで2分間反応させるだけで良く、ワークフローとしても合理的です(図2)。

逆転写酵素
逆転写酵素として知られている酵素群は、RNAをテンプレートとして相補的DNAを合成しますが、機能活性や性質といった点において違いがあります。そのような酵素間の性質の違いは、長鎖のRNA、GC含量の高いRNA、二次構造の複雑なRNA、そして品質の最適でないRNAなどを逆転写する能力に大きく影響します。
分子生物学において最も利用されているのは、トリ骨髄芽球症ウイルス(AMV)やモロニーマウス白血病ウイルス(MMLV)のpol遺伝子に由来する逆転写酵素です。AMV由来の逆転写酵素は、実験室でcDNAを合成するために分離された最初の酵素群の1つでした。本酵素は170 kDaのヘテロ二量体で、最適な反応温度は42~48°Cの間です。AMV由来の逆転写酵素は、RNA:cDNAハイブリッド中のRNA鎖を分解する強いRNase H活性を持っているため、短鎖のcDNAフラグメント(<5 kb)を生成します。
MMLV由来の逆転写酵素は、モノマー構造であることから、よりシンプルなクローニングや、組み換え体への改変が可能であり、代替品として好まれるようになりました。MMLV由来の逆転写酵素は75 kDaで、最適な反応温度は37°C付近です。AMV由来の逆転写酵素に比べると熱安定性は低いですが、本酵素はRNase H活性が低いため、より長鎖のcDNA(<7 kb)を合成することができます[3]。
cDNA合成をより改善するために、よりRNase H活性が低く(RNase Hドメインの変異、もしくはRNaseH、熱安定性が高く(55°Cまで)、そして処理能力を高めた(65倍以上)MMLV逆転写酵素の改変体が作られました。これらの性質は、cDNAの鎖長と収率の増加、感度の向上、阻害物質に対する耐性の改善、そして反応時間の短縮といった利点をもたらしました(表 2)[4]。(逆転写酵素の特性を参照してください)。
表 2.一般的な逆転写酵素とその特性
| AMV逆転写酵素 | MMLV逆転写酵素 | 改変型MMLV逆転写酵素 (Invitrogen™ SuperScript™ IV Reverse Transcriptase) | |
|---|---|---|---|
| RNase H活性 | 高 | 中 | 低 |
| 反応温度 (最も高い推奨温度) | 42°C | 37°C | 55°C |
| 反応時間 | 60分 | 60分 | 10 分 |
| ターゲットの鎖長 | ≤5 kb | ≤7 kb | ≤12 kb |
| 相対収量 (困難な条件あるいは最適でないRNAでの比較) | 中 | 低 | 高 |
プライマーの選択
逆転写酵素は逆転写を開始するにあたり、プライマーと呼ばれるDNAオリゴヌクレオチド(RNAテンプレート上の相補配列に結合し、新たな鎖合成の開始点としての役割を果たす)を必要とします。RNAテンプレートやアプリケーションに応じて、3種類の基本プライマー、すなわちオリゴ(dT)プライマー、ランダムプライマー、そして遺伝子特異的プライマーを使い分けます(図3)。

オリゴ (dT) プライマーは、真核生物のmRNA(トータルRNAの1~5%に相当)のポリ(A)テールにアニールする12~18個のデオキシチミジン残基で構成されます。このプライマーは、真核生物由来mRNAのcDNAライブラリの構築、完全長cDNAクローニング、そして3′ RACEに適しています。オリゴ(dT)プライマーは、ポリ(A)テールに特異的であるため、ホルマリン固定やパラフィン包埋(FFPE)試料から得られるような分解したRNA、そして原核生物由来RNAやマイクロRNAのようにポリ(A)テールのないRNAには適していません。オリゴ(dT)プライマーでは、cDNA合成が3′末端のポリ(A)テールから開始されるため、3′末端側への偏りを引き起こす可能性があります。著しい二次構造を持ったRNAもまた、完全長cDNAの合成を中断させることがあり、5′末端側の転写阻害を引き起こします。
逆転写反応の効率を改善するために、オリゴ(dT)プライマーを修飾することもできます。例えば、オリゴ(dT)プライマーの長さを20ヌクレオチドか、あるいは、より高温での逆転写反応においてそれらがアニーリングし得る長さであればもっと長く伸ばしても良いでしょう。場合によっては、オリゴ(dT)プライマーは、dN(dA、dT、dG、dC)やdV(dG、dA、dC)のような縮重塩基を3′末端に含んでも良いでしょう。このような修飾は、ポリ(A)スリップを防ぎ、ポリ(A)テールすぐ上流のプライミングサイトをロックします。このようなプライマーは、アンカーオリゴ(dT)と呼ばれます。
ランダムプライマーは、ランダムな塩基配列を持ったオリゴヌクレオチドです。このプライマーは、しばしば6個のヌクレオチドから成り、通常はランダムヘキサマーと呼ばれ、N6やdN6と標記されます。ランダムプライマーは、ランダムに結合する(つまりテンプレートの特異性がない)ため、試料中のいかなるRNA種ともアニールできる可能性を持ちます。そのため、このプライマーは、ポリ(A)テールのないRNA(例えば、rRNA、tRNA、ノンコーディングRNA、スモールRNA、原核生物のmRNAなど)や、分解したRNA(例えば、FFPE組織由来など)や、二次構造を持つことが知られているRNA(例えば、ウイルスゲノムなど)の逆転写反応に利用されます。
ランダムプライマーは検出を目的としたcDNA合成には向いているものの、長鎖RNAの完全長での逆転写には適していません。逆転写反応系内におけるランダムヘキサマーの濃度を上げればcDNAの収量は改善しますが、同じテンプレート上の複数サイトへの結合が増加することで、より短いcDNA断片が生じることになります(図4)。
そのうえ、ランダムプライマーは、幾つかのRT-PCRアプリケーションにとっては理想的ではありません。例えば、mRNAのコピー数が過剰に見積もられてしまうことが懸念されます[5]。オリゴ(dT)とランダムプライマーの混合物が、各々のプライマーの利点を活用する目的で、ツーステップRT-PCRにおいてしばしば用いられます。マイクロRNA(miRNA)発現アッセイには、ランダムヘキサマーは適しておらず、miRNAの逆転写には、特別なプライマーを設計しなければなりません[6,7]。

- 遺伝子特異的プライマーは、逆転写において最も特異的なプライミングを提供します。このプライマーは、ターゲットとなるRNAの既知の配列情報に基づいて設計されます。プライマーは特異的なRNA配列のみに結合するため、ターゲットRNAごとの遺伝子特異的プライマーのセットが必要になります。結果として、複数のターゲットRNAを分析するためには、より多くのRNAが必要になります。遺伝子特異的プライマーは通常、ワンステップのRT-PCRアプリケーションに使用されます。
表 3.一般的な逆転写反応用プライマーの比較
| オリゴ(dT) | ランダムヘキサマー | オリゴ(dT)+ ランダムヘキサマー | 遺伝子特異的プライマー | |
|---|---|---|---|---|
| 一般的な最終濃度 | 2~5 µM | 2~5 µM | 各1~2 µM | 0.5~1 µM |
| 25°Cでのプレプライマー伸長 | — | — | ||
| 主な利点 | ポリ(A)テールを持つRNAの完全長逆転写 | 分解したRNAを含めたほとんどのRNA種の逆転写 | オリゴ(dT)とランダムプライマーの利点を合わせ持つ | 目的の遺伝子に特異的な逆転写 |
逆転写反応の構成成分
逆転写反応の構成成分には、酵素とプライマーに加えて、RNAテンプレート(ゲノムDNA除去処理済みのもの)、緩衝液、dNTP、DTT、RNase阻害剤、そしてRNaseフリーの水が含まれます(図5)。

- RNAテンプレートは、前項で説明したように調製します。表 4は逆転写反応において推奨されるインプットRNA量の範囲を示しています。最適な量はターゲット配列の存在率と逆転写酵素の感度に依存します。
表 4.逆転写反応において推奨されるインプットRNA量の範囲
| RNAテンプレート | 推奨される範囲 |
|---|---|
| トータルRNA | 10 pg~5 μg |
| ポリ(A)RNA | 10 pg~5 μg |
| 特定のRNA | 0.01 pg~5 μg |
- 反応緩衝液は反応に好ましいpHとイオン強度を維持します。キットなどで提供される緩衝液には、逆転写反応の効率を増強させるための添加剤が含まれる場合があります。
- dNTPは通常、各0.5~1 mMとし、なるべく等モル濃度にしておきます。逆転写反応を確実なものとするため、希釈してすぐの品質の良いdNTPの使用をお勧めします。
- 還元剤であるDTTは通常、各0.5~1 mMとし、なるべく等モル濃度にしておきます。DTTや他の添加剤が沈殿する場合、反応効率が落ちることがありますので、反応成分はしっかりと混合し、十分に溶解してください。
- RNase阻害剤はRNAの分解を抑えるために、通常、反応緩衝液に含まれているか、あるいは反応系に添加します。RNaseは分離の過程で共精製されるか、あるいは反応のセットアップの段階で混入することがあります。多くのRNaseの存在が知られているため、適したRNase阻害剤を、酵素の作用機序と反応条件に基づいて選択する必要があります。
- 水ヌクレアーゼフリーのものを使用します。市販されているヌクレアーゼフリーの水か、あるいは、DEPC (diethylpyrocarbonate)処理された水の使用がベストです。混入したRNaseは簡単なろ過では除去できず、また熱に安定であることから、オートクレーブした水では十分ではありません。
反応温度と反応時間
逆転写反応は3つのメインステップ、すなわち、プライマーのアニーリング、DNAの重合化、そして酵素の不活化をともないます(図6)。これらのステップの温度と時間は、プライマーの選択、ターゲットとなるRNA、そして使用する逆転写酵素によって変わります。

- プライマーのアニーリング:RNAテンプレートとプライマーを混合し、65°Cで5分間加熱した後、氷上で少なくとも1分間インキュベートします。この操作によって、RNAは一本鎖となり、プライマーは標的に効率良くアニールします。アニーリング後に、逆転写酵素や必要な成分(緩衝液、各dNTP、RNase阻害剤など)を加えます。
- DNAの重合化:このステップにおける反応温度と反応時間は、選択したプライマーと使用する逆転写酵素によって変わってきます。オリゴ(dT)プライマー(Tm約35~50°C)の場合、逆転写酵素の最適温度(37~50°C)で直接インキュベートできます。ランダムヘキサマーは鎖長が短いため、大抵は低いTm値(約10~15°C)を持ちます。そのため、ランダムヘキサマーを(単独で、あるいはオリゴ(dT)と組み合わせて)使用する場合、プライマー伸長のために酵素を加えた後、室温(約25°C)にて10分間、反応液をインキュベートすることをお勧めします。
逆転写酵素はそれぞれに耐熱性が異なり、最も高い最適反応温度が決まっています。耐熱性の逆転写酵素を使えば、より高い温度(50°Cなど)での反応が可能であるため、酵素活性に悪影響なく、GC含量の高いRNAや二次構造を持ったRNAを変性させることができます(図 7)。そのような酵素による、高温でのインキュベーションは、cDNAの収量や鎖長を増加させ、遺伝子産物の反映を向上させます。
重合化の時間は逆転写酵素の処理能力に依存し、処理能力とは基質との結合1回あたりに組み込まれるヌクレオチドの数で定義されます。例えば、処理能力の低い野生型のMMLV逆転写酵素では、cDNAの合成に60分以上を要します。それに対して、処理能力の高い改変型の逆転写酵素では、9 kbのcDNAを合成するのに10分程度しかかかりません(詳細については逆転写酵素の処理能力を参照)。

- 酵素の不活化逆転写における最後のステップは、逆転写酵素の不活化です。不活化は酵素の耐熱性に応じて、70~85°Cの範囲で行われます。不活化は通常5~15分の間ですが、できる限り高温で短時間行います。
第一鎖および第二鎖cDNAの合成
前項でも述べたように、RNAテンプレートによるcDNAの合成では、cDNA:RNAハイブリッドが生成されます。このプロセスは、第一鎖cDNAの合成と呼ばれています。RNase H活性が存在すれば(野生型のAMVやMMLV逆転写酵素のように)、cDNA:RNAハイブリッドのRNA鎖は、第一鎖合成の間に分解されます。第一鎖cDNA(アニールしたRNAの有無に関わらず)は、RT-PCRのようなアプリケーションに直接使用でき、その際、耐熱性のDNAポリメラーゼ(例えばTaqDNAポリメラーゼなど)がcDNAの相補鎖を複製します。
cDNAライブラリの構築やシーケンシングでは、第一鎖cDNAは、ターゲットRNAを反映する2本鎖cDNAを生成するためのテンプレートとして利用されます。このプロセスは、第二鎖cDNAの合成と呼ばれています。第二鎖cDNAの合成では、cDNAの長さと収量を最大化するため、最小限のRNase H活性を持つ逆転写酵素が推奨されます。
2本鎖cDNAの合成では、しばしば異なるDNAポリメラーゼ(例えば、T7 DNAポリメラーゼ、DNAポリメラーゼI、TaqDNAポリメラーゼなど)が、第一鎖cDNAの相補鎖を合成するために用いられます。Gubler and Hoffmanの方法として以下に示すように、2本鎖cDNAの合成では追加の酵素を添加します[8] (図8)。

- E. coliRNase Hは、cDNA:RNA複合体のRNA鎖を刻み込み、DNA合成のための3′-OHプライミングサイトを生成します。
- 大腸菌DNA ポリメラーゼ I は、5′3′ポリメラーゼ活性によって刻まれたRNA鎖を伸長し、5′3′エキソヌクレアーゼ活性によって合成方向にRNA鎖を置き変えます。
- E. coliDNAリガーゼは、新たに合成されたcDNAセグメント間の切れ目を閉じます。(T4 DNAリガーゼは、平滑末端を持つ2本鎖cDNAフラグメントをライゲートし、キメラ構造を形成するため、お勧めしません)
- T4 DNA ポリメラーゼは、2本鎖cDNAの末端を平滑にします(最終ステップにおけるオプション)。
結論として、逆転写の成功の可否は、反応成分と反応条件に強く依存しています。目的とするダウンストリームのアプリケーションに適合するように、反応を実行してください。
参考文献
- Schroeder A, Mueller O, Stocker S et al.(2006) The RIN: an RNA integrity number for assigning integrity values to RNA measurements.BMC Mol Biol7:3.
- Mueller O, Lightfoot S, Schroeder A (2016) RNA Integrity Number (RIN) – Standardization of RNA Quality Control.Agilent Technologies Publication 5989-1165EN.
- Invitrogen Corp. (2002) High performance RT for reliability in every experiment.(Brochure)
- Thermo Fisher Scientific (2015) SuperScript IV Reverse Transcriptase.(White paper)
- Zhang J, Byrne CD (1999) Differential priming of RNA templates during cDNA synthesis markedly affects both accuracy and reproducibility of quantitative competitive reverse-transcriptase PCR.Biochem J337 (Pt 2):231–241.
- Chen C, Ridzon DA, Broomer AJ et al.(2005) Real-time quantification of microRNAs by stem-loop RT-PCR.Nucleic Acids Res33(20):e179.
- Kramer MF (2011) Stem-loop RT-qPCR for miRNAs.Curr Protoc Mol BiolUnit 15.10.
- Gubler U, Hoffman BJ (1983) A simple and very efficient method for generating cDNA libraries.Gene25(2-3):263–269.
For Research Use Only. Not for use in diagnostic procedures.